 欢迎来到乐鱼网页版登录入口网站!
欢迎来到乐鱼网页版登录入口网站! 欢迎来到乐鱼网页版登录入口网站!
欢迎来到乐鱼网页版登录入口网站!
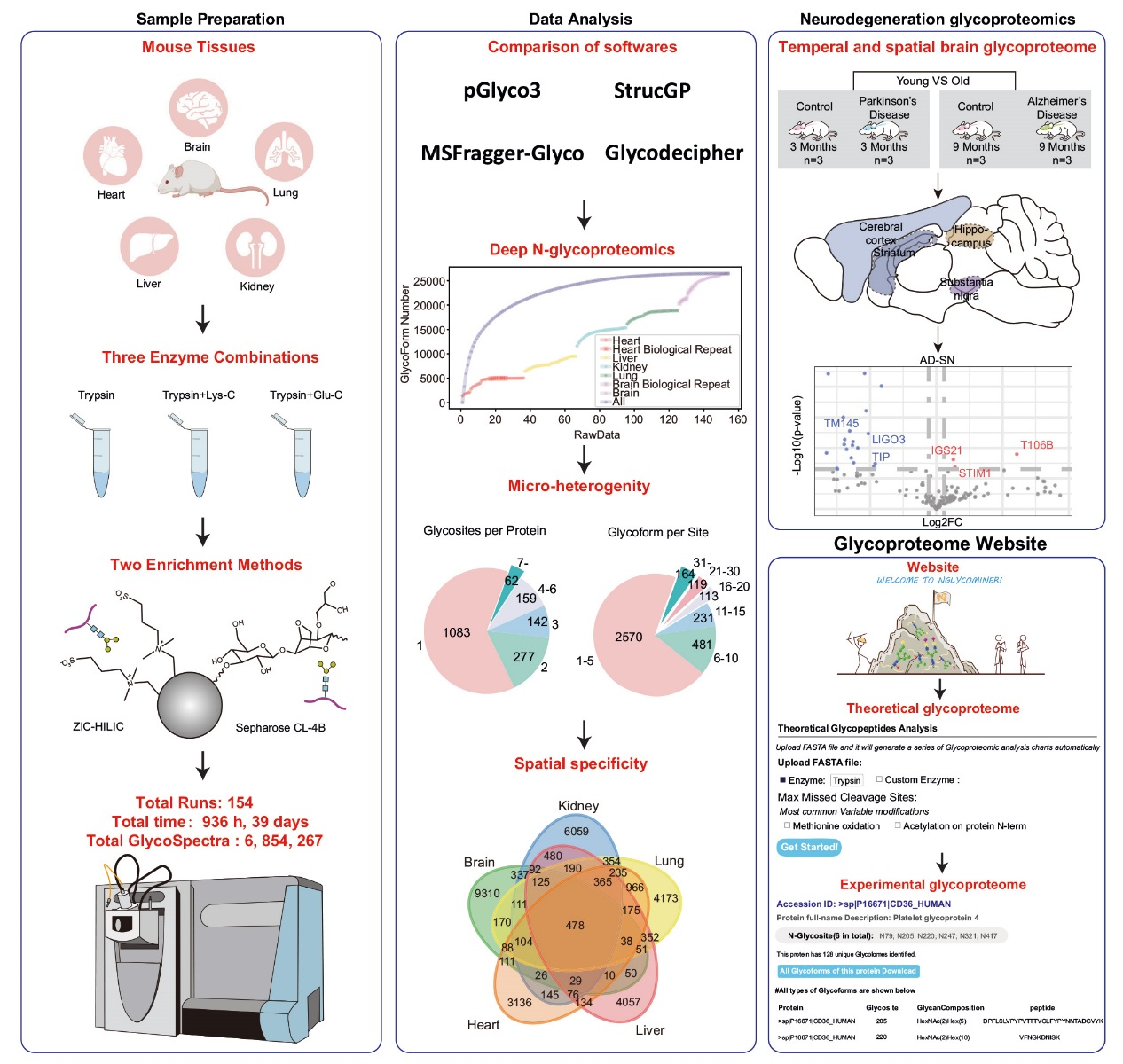
构建迄今规模最大的小鼠多器官N -糖蛋白组数据库。聚焦小鼠四个关键脑区开展时空维度糖蛋白组学研究,揭示糖基化在衰老及神经退行性疾病中的变化规律,并搭建在线数据库 NGlycoMiner,为相关研究提供数据支持。
《Nature Communications》(简称 “Nat Commun”)是自然科研(Nature Research)旗下的国际顶级开放获取(Open Access)综合性学术期刊,2010年正式创刊,旨在发表自然科学领域(涵盖生物、化学、物理、地球科学、医学等)具有重要科学意义、但未达到《Nature》主刊突破性高度的原创研究成果,填补了顶级主刊与专业子刊间的发表空白。
出版周期: Bimonthly;
影响因子:2024-2025最新影响因子为15.7,五年影响因子为17.2;
ISSN:2041-1723;
发文量:2024 年发文量为10749篇;
版面费:$6790.00/篇;
一、研究背景
蛋白质糖基化作为广泛存在的关键蛋白质翻译后修饰,其聚糖结构具有多样性、复杂性与动态性,对蛋白质功能影响深远,在生理与病理过程中均发挥核心作用。N-糖蛋白由聚糖连接多肽链特定天冬酰胺残基形成,其合成受多种因素调控,识别糖蛋白并表征位点特异性聚糖,对解析健康与疾病机制至关重要。但基于质谱的N-糖蛋白质组学面临诸多挑战,如异质性、糖肽丰度低、富集不完全,且质谱图质量差、复杂,导致光谱识别率低、假发现率高。虽科研人员优化样品制备、改进技术与开发软件,仍未达理想识别深度与精度。此前用pGlyco 2.0构建的小鼠数据集,已无法满足当前AI算法对糖肽光谱研究的高质量训练数据需求,成为领域瓶颈。
二、关键技术总结
样本处理:选取小鼠多种组织(脑、肺、肾、肝、心脏)及疾病模型脑区样本,采用含蛋白酶抑制剂的裂解缓冲液匀浆组织,经DTT还原、IAA烷基化、丙酮沉淀处理蛋白质,再用胰蛋白酶单独消化或与Lys-C、Glu-C组合消化蛋白质,最后通过Sep-Pak C18柱脱盐。
糖肽富集:采用ZIC-HILIC和Sepharose CL-4B两种方法。ZIC-HILIC法将肽段加载到含 ZIC-HILIC介质的微柱,经多步洗涤后梯度洗脱;Sepharose CL-4B法让肽段与介质振荡结合,洗涤后孵育回收糖肽。
液相色谱-质谱分析:常规组织样本用Orbitrap Fusion质谱仪结合Proxeon EASY-nLC II液相色谱泵,以特定流动相和梯度洗脱;疾病模型脑区样本用配备FAIMSpro接口的Orbitrap Exploris 480质谱仪与Easy-nLC 1200系统联用,采用不同梯度洗脱,均通过HCD fragmentation获取MS/MS数据。
数据处理与分析:从UniProt下载小鼠蛋白质数据库,用pGlyco3、StrucGP、MSFragger-Glyco、Glyco-Decipher四种软件进行数据库搜索;基于XIC面积定量,采用总强度归一化处理数据;通过Python和R进行生物信息学分析,包括二级结构分布分析、PCA、层次聚类、Pearson相关分析、GO和KEGG通路富集分析,以及WGCNA构建共调控网络。
验证与数据库构建:用Western blot 分析验证糖蛋白表达,通过PNGase F处理去除N - 糖链辅助验证;基于Django Web框架、MySQL数据库、Python后端及HTML/CSS/JavaScript 前端,结合Nginx和uWSGI构建N-GlycoMiner 数据库,整合实验数据与文献数据。
三、主要研究成果
选取了五种小鼠组织进行深度分析,包括脑、肺、肾、肝和心脏;使用了三种酶解方案以最大化肽段覆盖度:Trypsin(胰蛋白酶)、Trypsin + Lys-C、Trypsin + Glu-C;Trypsin + Glu-C;采用ZIC-HILIC(亲水相互作用色谱)和Sepharose CL-4B(基于凝集素的富集)两种糖肽富集策略以捕获更广泛的糖肽;共进行了154次 LC-MS/MS运行,总耗时 936小时(39天),最终获得了685万张 包含氧鎓离子的糖肽质谱图(Glyco-spectra)。
使用四款主流糖蛋白组学软件(pGlyco3, StrucGP, MSFragger-Glyco, Glyco-Decipher)对数据进行联合搜索和鉴定,以评估各软件性能并提高鉴定可信度。展示了不同组织中鉴定到的糖肽数量,证明了数据集的深度。分析了同一糖基化位点上连接不同聚糖的现象。通过图表对比,直观显示了糖基化模式在不同组织间的显著差异。
对阿尔茨海默病(AD)、帕金森病(PD)、衰老和年轻对照组的小鼠海马体、前额叶皮层、纹状体、和黑质进行了时空分析。旨在揭示糖基化在脑老化与神经退行性疾病中的时空特异性变化。
构建了一个名为 N-GlycoMiner 的在线数据库平台(www.NGlycoMiner.com)。用户可查询本研究中所有鉴定到的糖蛋白、糖基化位点和糖型的详细信息。网站提供理论糖基化分析功能,用户可上传FASTA文件,自动预测潜在的N-糖基化位点和生成模拟糖肽。整合了AlphaFold2预测的蛋白结构,并在结构上标注已鉴定的糖基化位点。
核心结果是通过一个极其全面和深入的工作流程,整合了多维度的实验设计、多软件的数据分析、疾病时空模型以及一个强大的数据库资源,成功构建了迄今为止最大、最深度的小鼠N-糖蛋白组图谱,为揭示糖基化在生物学中的复杂作用提供了强大的数据基础和研究平台。
2、多软件鉴定结果的综合评估与数据质量验证
鉴定数量差异:四款软件(pGlyco3, StructGP, MSFragger-Glyco, Glyco-Decipher)共鉴定出约104万个糖肽谱图匹配(GPSMs),但各软件鉴定数量存在显著差异。在前体、糖型层次上,各软件鉴定数量排名一致(Glyco-Decipher > MSFragger-Glyco > pGlyco3 > StructGP);但在糖基化位点、糖蛋白层次上,排名出现变化,揭示了不同软件的设计偏好和局限性。仅有 160,928个GPSMs(占总GPSMs的15.5%) 被所有四款软件一致鉴定为相同的糖肽前体,被视为高可信度数据。基于软件间的一致性,提出了一个四级可信度系统:
高可信度 (High-confidence): 四款软件一致 (15.5%);中可信度 (Moderate-confidence): 至少两款软件一致 (37.7%);低可信度 (Low-confidence): 仅一款软件鉴定 (38.7%);模糊鉴定 (Ambiguous): 不同软件给出完全不同结果 (8.1%);其中,pGlyco3表现出最高的一致性(最可靠),而MSFragger-Glyco虽然灵敏度高(鉴定数量多),但与其他软件的不一致性也最高。
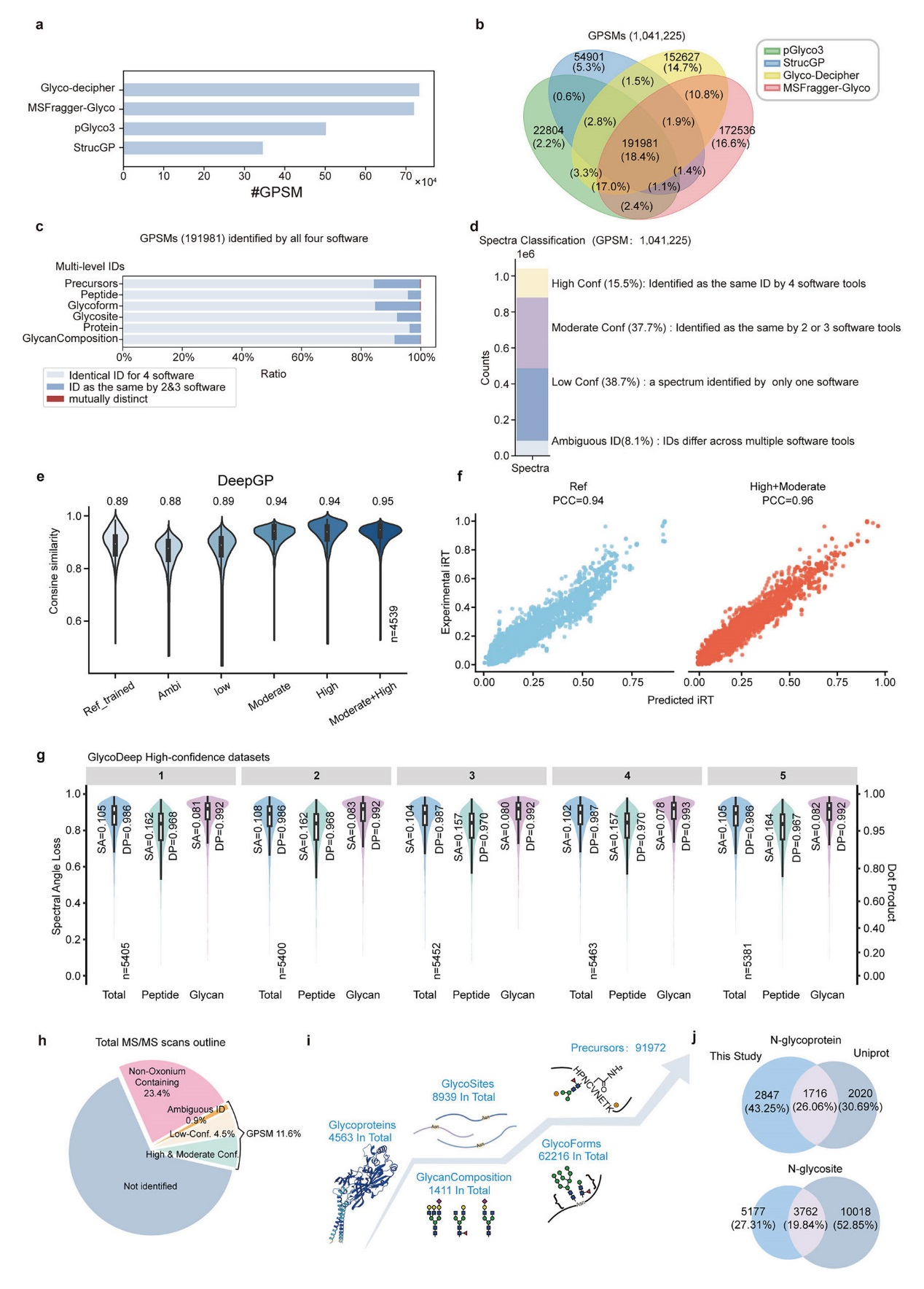
图2、多软件鉴定综合分析结果
使用不同可信度的数据集重新训练了DeepGP和DeepGlyco等AI模型,使用中高可信度数据合并训练的模型,其预测谱图与实验谱图的余弦相似度中位数高达0.95,性能优于文献中已报道的模型,使用中高可信度数据训练的模型,在保留时间(IRT)预测上也表现出更高的准确性(皮尔逊相关系数更高),5折交叉验证表明,基于高可信度数据训练的DeepGlyco模型预测结果极其稳定且准确(点积中位数>0.986)。
去除模糊鉴定后,最终构建的数据集包含:91,972个 唯一前体糖肽,62,216个唯一糖型,8,939个糖基化位点,4,563个糖蛋白;本研究鉴定到的糖蛋白和糖基化位点数量远超UniProt数据库中的记录,分别多出2,847个糖蛋白和5,177个位点,极大地扩展了已知的小鼠N-糖蛋白组图谱。尽管投入巨大,但总谱图的鉴定率仅为11.6%,仍有88.4%的谱图未被鉴定,凸显了当前糖蛋白组学技术在谱图解析能力上的巨大挑战和未来改进空间。
研究通过多软件交叉验证,构建了一个具有可信度分级的、超大规模且高质量的小鼠N-糖蛋白组数据集。该数据集不仅本身规模空前,而且能显著提升AI模型的预测性能,为糖蛋白组学领域的算法开发和生物学发现提供了宝贵的资源。同时,结果也客观地揭示了当前技术仍存在解析度不足的局限性。
3、小鼠N-糖蛋白组的综合分析
质谱分布分析结果表明,完整糖肽分子量主要分布在 2000-6000 Da 范围内,而去糖基化肽段质量多在 1000-2500 Da 之间,与理论预测(所有含N-X-S/T/C序列的肽段)的分布相比,揭示了质谱技术在可检测质量范围上的局限性。脑组织中的聚糖整体上明显小于其他组织,这表明大脑拥有独特的糖基化谱,提示其糖基化功能可能与其他器官不同。利用AlphaFold2和DSSP分析了糖蛋白的二级结构。结果显示,N-糖基化位点更多地位于Coil(无规卷曲)和 Bend(弯曲)区域,其次是 β-strand(β-折叠) 和 Turn(转角) 区域。构建了三维气泡图来可视化糖蛋白的异质性,三个维度分别为:X轴,糖蛋白;Y轴,每个蛋白上的糖基化位点数量;Z轴,每个蛋白上的糖型总数;平均每个蛋白有 ~2个 糖基化位点,每个位点有 ~7种 不同的糖型(微观异质性)。脑组织的糖蛋白表现出最高的微异质性,而心脏组织的最低。某些蛋白在不同组织中表现出截然不同的糖基化模式,如CD36(血小板糖蛋白4)在大脑中仅检测到1个糖基化位点(N417)和2种糖型。而在心脏和肺中检测到全部7个 理论位点,其中心脏中有多达 258种 糖型。蛋白质印迹(Western Blot)验证实验结果与质谱数据一致,心脏和肺中糖基化CD36的蛋白表达量远高于大脑。使用PNGase F酶去除N-糖链后,条带发生迁移,证实了CD36的修饰主要是N-糖基化。这表明,不同组织间CD36糖基化水平的差异主要源于其蛋白表达水平本身的高低。CD36在心脏和脂肪组织中负责脂肪酸摄取,并与肺癌发展有关。其糖基化的组织特异性暗示了糖基化对于调控CD36在不同组织中执行特定功能至关重要。
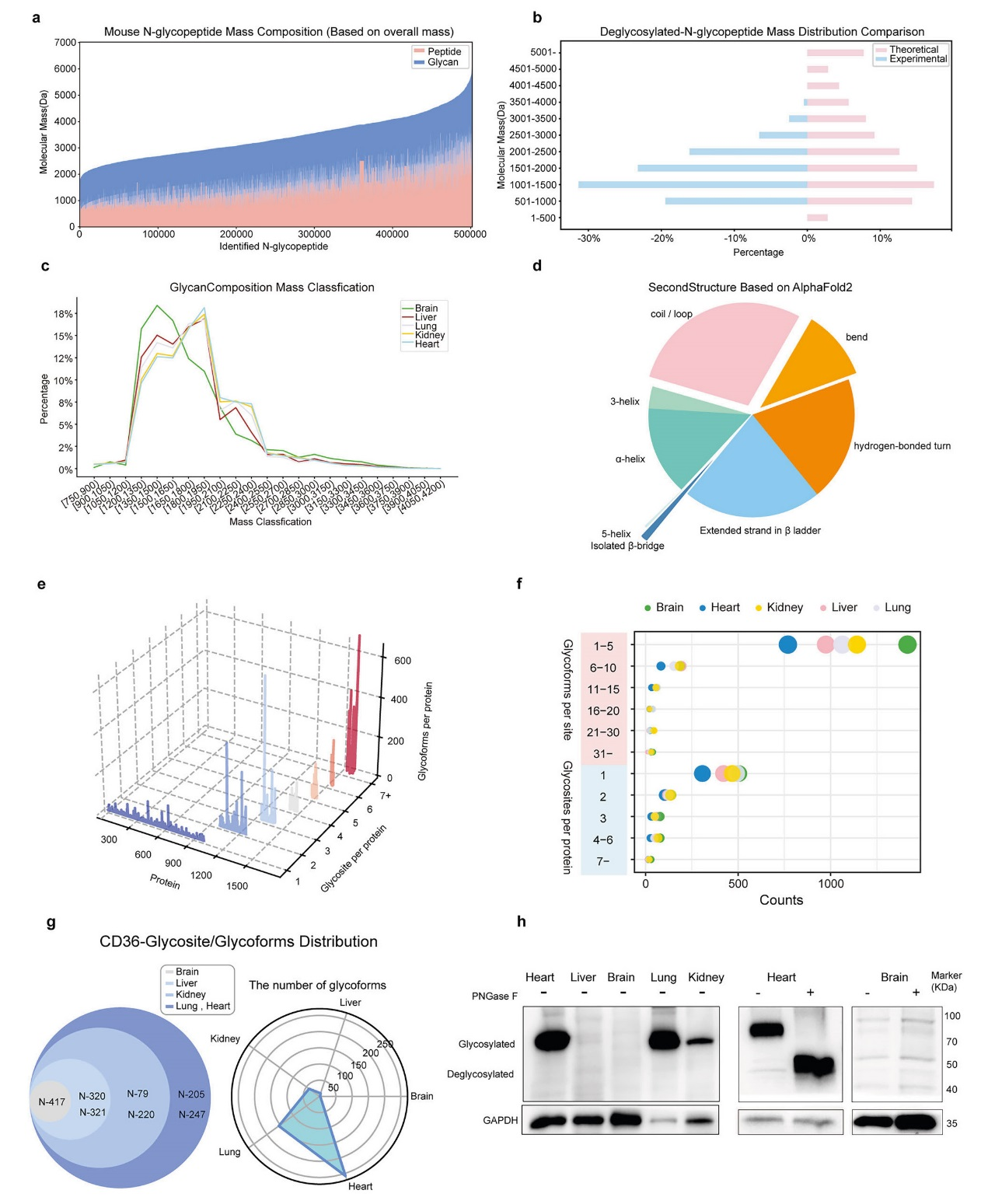
图3、小鼠N-糖蛋白组的综合分析结果
研究利用超深度数据集,全面描绘了小鼠N-糖蛋白组的整体特征,揭示了糖基化修饰在分子大小、蛋白结构偏好和异质性程度上的规律。最重要的是,它通过令人信服的数据(包括对CD36的生化验证)证实了糖基化具有显著的组织特异性,这种特异性不仅体现在糖型种类上,更与底层蛋白的表达水平和器官的功能需求密切相关。这部分分析为后续探索大脑等特定器官在衰老和疾病中的糖基化变化奠定了坚实的基础。
系统揭示了小鼠五种组织中N-糖基化的高度特异性及其功能关联。主成分分析显示,脑组织的糖基化谱与其他组织截然不同,肾脏也展现出独特的聚糖模式。无监督聚类识别出四个聚糖表达簇:脑富集簇以岩藻糖化和NeuAc唾液酸化修饰为特征;心/肝簇富含NeuGc唾液酸;肾簇高岩藻糖但低唾液酸。对应地,糖蛋白表达谱聚类出五大组织特异性簇,其功能与器官生理完美契合:脑特异性糖蛋白主导神经发育与突触功能;肝、心、肾和肺的糖蛋白分别富集于代谢、机体稳态、物质运输和结构发育过程。共调控网络分析进一步发现,跨组织保守的糖蛋白显著参与ECM-受体相互作用、鞘脂代谢等通路,并鉴定出β-己糖胺酶亚基α(Hexa)为核心枢纽分子。这些结果表明,糖基化修饰并非随机,而是精确调控并支撑着组织的特异性功能。
研究通过多维度生物信息学分析,将糖基化数据与生物学功能直接联系起来。它系统地证明了N-糖基化修饰具有高度的组织特异性,这种特异性不仅体现在聚糖和糖蛋白的表达量上,更与其所在组织的核心生理功能完美契合(如神经功能、代谢、运输等)。同时,网络分析揭示了跨组织保守的糖基化调控通路和核心分子(如Hexa),为理解糖基化在系统生物学中的调控作用提供了新视角。
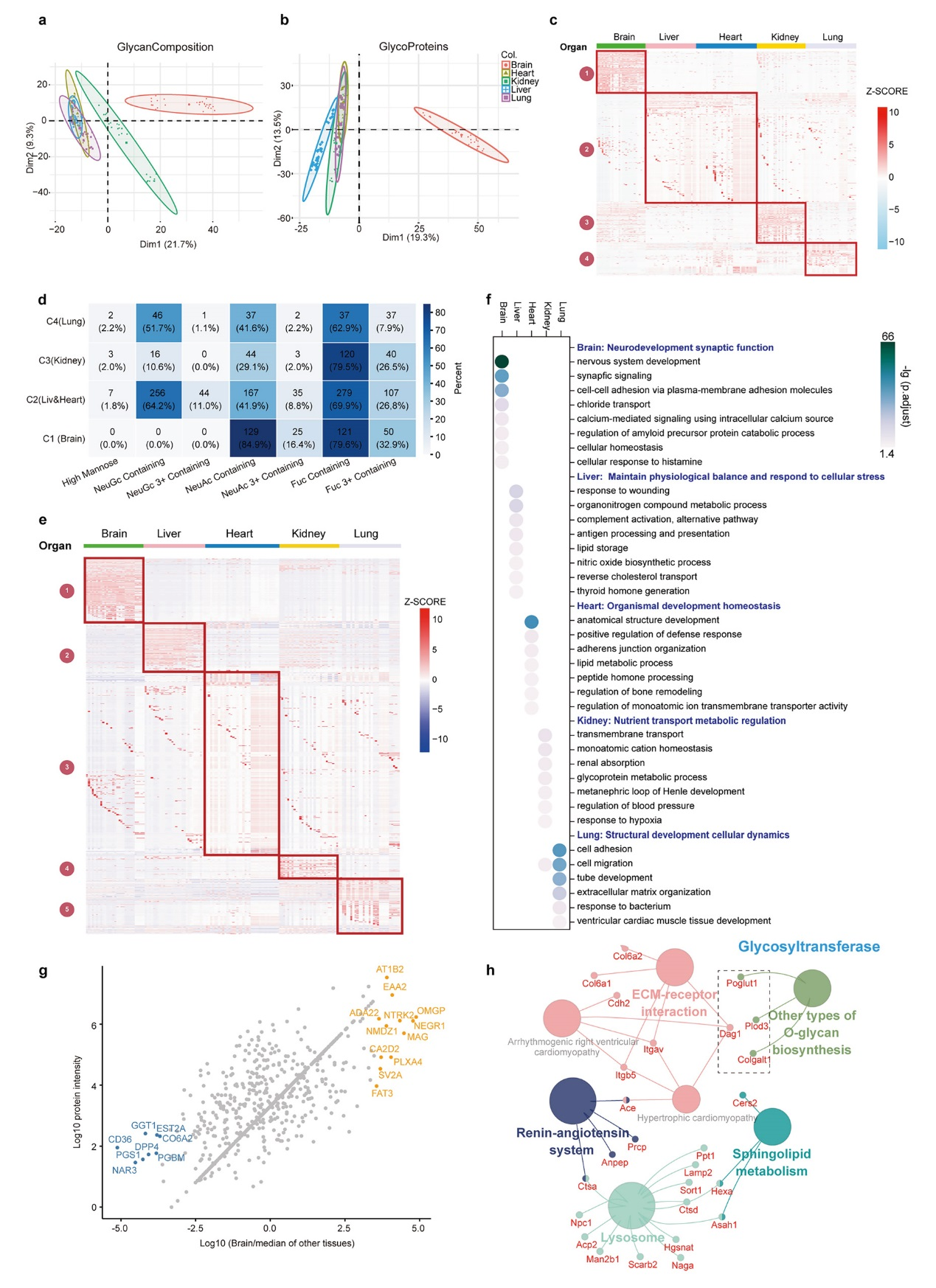
图4、小鼠跨组织的聚糖组成与糖蛋白的综合分析结果
研究分析了小鼠大脑N-糖基化在衰老与神经退行性疾病中的时空动态变化。结果表明,年龄是驱动糖基化变化的首要因素,其影响远超疾病状态,老年组(9个月)相比年轻组(3个月)多个脑区普遍出现糖基化水平下降。研究发现了显著的脑区与疾病特异性:阿尔茨海默病(AD)、帕金森病(PD)和衰老过程在不同脑区(如海马、纹状体、黑质)均引发独特的糖基化修饰改变。共表达网络分析(WGCNA)进一步识别出多个功能协同的糖基化模块,例如:模块M5(与轴突发生相关)在PD中上调;模块M6(与神经发育相关)在AD和衰老中下调;模块M8(溶酶体功能)在AD中上调。聚糖模块分析同样发现,高度唾液酸化的聚糖在AD和PD中均显著减少。

研究不仅证实了脑部N-糖基化具有强烈的区域特异性,更重要的是揭示了年龄是驱动其变化的最强因素,其影响甚至超过疾病本身。通过共表达网络分析,研究发现了多个与特定脑区、衰老及神经退行性疾病(AD, PD)密切相关的糖基化功能模块,并将这些变化与诸如突触功能、细胞粘附和溶酶体过程等关键生物学通路联系起来。这为理解糖基化在脑老化及神经退行性疾病中的分子机制提供了前所未有的时空动态视角和大量潜在的调控靶点。
6、N-GlycoMiner数据库的构建和功能
研究构建了综合性N-糖蛋白组学资源平台 - N-GlycoMiner (www.NGlycoMiner.com)。平台整合了本研究产生的大规模实验数据与近十年60篇文献中的公共数据,构建了迄今最全面的物种特异性N-糖蛋白组数据库。其核心内容包含:小鼠数据(31万种糖型、1.2万种糖蛋白、3.8万个糖基化位点)和人类数据(10.7万种糖型、8007种糖蛋白、1.7万个位点)。平台提供四大功能:1)本研究数据查询,可检索组织特异性表达、鉴定可信度等详细信息;2)文献数据整合,涵盖多种样本类型与疾病模型;3)生物学见解,聚焦神经疾病、癌症等病理中失调的糖基化模式;4)理论预测功能,支持用户上传蛋白序列,自动预测糖基化位点、生成模拟糖肽并分析其理化性质。该平台代表了当前该领域最全面的数据资源,旨在成为糖生物学研究领域的核心工具,为揭示糖基化在生理和疾病中的功能提供不可或缺的数据基础。
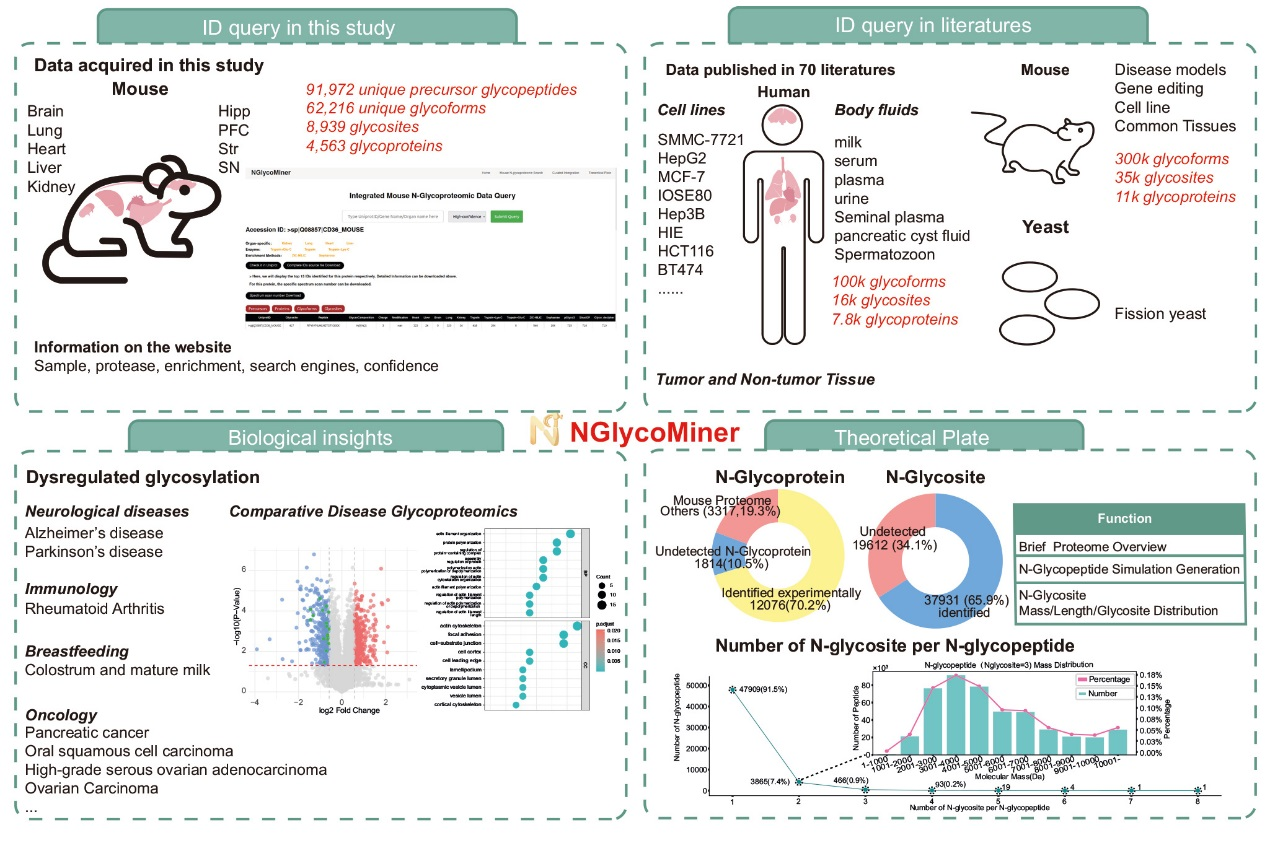
图5、N-GlycoMiner (www.NGlycoMiner.com) 的核心架构与功能展示
四、研究的意义
研究通过技术上的极致创新,产生了前所未有深度和高质量的数据,并以此为基础构建了支撑未来研究的平台资源,最终揭示了糖基化在生理和病理状态下前所未有的复杂性和功能性。这不仅极大地推动了糖蛋白组学领域本身的发展,更为神经科学、衰老研究和精准医学等多个相关领域提供了强大的新工具和深刻的新见解,具有里程碑意义。
参考文献:
Fang P, Yu X, Ding M, Qifei C, Jiang H, Shi Q, Zhao W, Zheng W, Li Y, Ling Z, Kong WJ, Yang P, Shen H. Ultradeep N-glycoproteome atlas of mouse reveals spatiotemporal signatures of brain aging and neurodegenerative diseases. Nat Commun. 2025 Jul 1;16(1):5568. doi: 10.1038/s41467-025-60437-6. PMID: 40593524; PMCID: PMC12215503.